专为研发设计的软件触手可及您的研发值得拥有的科学软件,该软件将提供见解,提高协作,提高效率并帮助您更快地创新
我们的软件提供
产品
是时候让您的软件支持您的科学
科学研究有独特的软件需求,但是研发团队经常发现自己在内部解决方案或使用不像化学家那样认为的软件来制作。
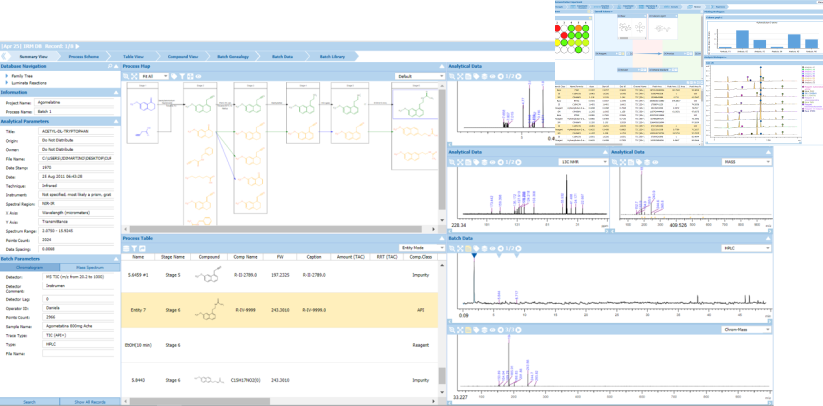
ACD/Labs软件设计有科学任务和工作流程,因此您可以…
- 传达您的科学
- 流线和自动化复杂工作流程
- 了解分子行为
- 支持HTE&CMC的决策
- 从分析数据中提取答案
- 分享知识与增强协作
寻找一个结构绘图软件来支持您的学习?
我们为个人和教育用途提供免费的chemsketch免费软件。
顾客评论
> 4,600
研发组织选择ACD/LABS软件
> 90%
我们的客户选择继续年复一年地使用我们的软件
> 85%
在全球前50名制药/生物制药和生物技术公司中,有我们的软件
8/10
全球顶尖的大学为学生和研究人员配备了我们的软件